Una panoramica sulle principali vie biochimiche della produzione di metano nei ruminanti.
Riassunto: I carboidrati (es. amido e cellulosa) sono la principale fonte di energia nella dieta delle vacche da latte. La digestione ruminale di amido e cellulosa è effettuata da microrganismi ed enzimi digestivi. Durante la degradazione anaerobica della parte fibrosa della razione, viene prodotta una notevole quantità di idrogeno, nocivo per il microbiota, che viene successivamente trasformato in metano grazie all’azione degli Archea. Attraverso l’eruttazione il metano prodotto viene rilasciato nell’atmosfera dando luogo non solo a una perdita di energia per l’animale, ma anche ad un problema ambientale di entità non trascurabile. Allo scopo di ridurre la formazione di metano è assolutamente necessario comprendere il processo chimico che porta alla formazione di questo gas serra ed identificare i microrganismi e gli enzimi coinvolti per conoscerne il ruolo e l’attività. Accanto alle tecniche analitiche esistenti vengono continuamente sviluppate nuove tecniche che stanno permettendo una veloce crescita della nostra conoscenza della comunità microbica amilolitica e cellulolitica nel rumine delle vacche da latte. Utilizzando metodi tradizionali basati su colture, sono stati isolati i principali batteri, funghi e protozoi amilolitici e cellulolitici nel rumine delle vacche da latte. È stato scoperto che questi microbi coltivabili rappresentano solo una piccola frazione della popolazione totale di microrganismi presenti nel rumine. Un’applicazione più recente dell’approccio della metagenomica indipendente dalla coltura, ci ha permesso di acquisire una struttura genetica più completa e una composizione funzionale della comunità microbica del rumine.
Introduzione
L’allevamento dei ruminanti da latte o da carne è una parte fondamentale dell’economia mondiale e la comprensione del complesso ecosistema microbico ruminale è fondamentale per ottimizzare la produttività e la salute degli animali, nonché limitare l’impatto ambientale degli allevamenti. L’alimentazione dei ruminanti è composta da un’ampia gamma di materie prime di origine vegetale, che sono scomposte nel prestomaco dei ruminanti (reticolo-rumine) e fermentate in acidi grassi volatili (VFA), anidride carbonica (CO2) e metano (CH4). Il metano è prodotto da un gruppo di microrganismi chiamati Archaea metanogenici, utilizzando una gamma limitata di substrati principalmente nel rumine, e viene rilasciato nell’atmosfera tramite eruttazione, con solo il 10% di CH4 prodotto dalla fermentazione nell’intestino posteriore. Le emissioni dei ruminanti sono state collegate al riscaldamento globale e ai cambiamenti climatici, e c’è un crescente interesse nel trovare modi per ridurle, e diminuire la perdita di energia dall’animale e l’impatto ambientale dei ruminanti. Di fondamentale importanza è quindi identificare i microbi e gli enzimi coinvolti nella degradazione dell’amido e della cellulosa, conoscerne il ruolo e l’attività, e comprendere chiaramente quali sono le principali vie biochimiche della metanogenesi nei ruminanti.
Archaea nel rumine
La dieta delle vacche da latte è composta principalmente da cereali ricchi di amido e da cellulosa che rappresentano il 20-40% della razione. L’amido è un polisaccaride caratterizzato da un gran numero di unità di glucosio polimerizzate unite tra loro da legame α-glicosidico e costituito da 4/5 di amilopectina e da 1/5 di amilosio. In entrambi i casi si tratta di polimeri del glucosio che si differenziano l’uno dall’altro per la struttura. L’amilosio è un polimero lineare che tende ad avvolgersi ad elica, in cui le unità di glucosio sono legate tra loro con legami glicosidici α(1→4) (tra il sito 1 di una unità e quello 4 dell’unità successiva). L’elica è costituita da 6 molecole di glucopiranosio per spira, stabilizzate da legami a idrogeno come nel DNA. L’amilopectina è invece un polimero ramificato che presenta catene di base di struttura simile all’amilosio che si dispongono a formare una struttura ramificata; ogni 24-30 unità di glucosio si innestano catene laterali attraverso legami α(1→6). L’amilopectina ha una struttura a grappolo costituita da segmenti di tipo A (15-20 unità di glucopiranosio) e catene di tipo B meno numerose. Sullo scheletro delle catene di Tipo B si insediano le catene di tipo A che costituiscono le ramificazioni. Una molecola di amilosio può contenere sino a 1000 residui di glucosio. L’amilosio lega insieme la struttura più espansa dell’amilopectina.
La cellulosa è un polisaccaride composto da numerose unità di glucosio collegate tra loro formando la catena lunga. Le catene sono disposte parallelamente le une alle altre e si legano fra loro per mezzo di legami ad idrogeno, formando fibrille, catene molto lunghe, difficili da dissolvere. Il legame tra l’unità di glucosio o legame glicosidico è β (1-4). La catena non è ramificata ma lineare e contiene da 10.000 a 15.000 unità di D-glucosio.
Nell’intestino dell’uomo il processo di idrolizzazione della cellulosa con trasformazione del legame beta in alfa non avviene perché mancano gli enzimi per rompere il legame β(1→4) glicosidico. Nei prestomaci dei ruminanti e nell’intestino cieco degli erbivori monogastrici (equidi), invece, sono presenti numerosi batteri e protozoi simbionti che convertono il legame β in un legame α, scindibile da tutti gli animali. I ruminanti sono caratterizzati dall’avere un apparato digerente molto complesso, formato da tre prestomaci (rumine, reticolo, omaso) e da uno stomaco vero e proprio (abomaso). La digestione avviene nell’abomaso ed è preceduta da una fermentazione microbica, che avviene nei prestomaci. La degradazione dell’amido avviene principalmente nel rumine, in parte nell’intestino tenue ed infine nell’intestino posteriore dei ruminanti.
Il rumine può essere quindi considerato un fermentatore popolato da miliardi di microrganismi, responsabili delle trasformazioni della sostanza organica proveniente dalla dieta. La comunità microbica nel suo complesso comprende i batteri, gli archea, i protozoi ed i funghi. Ciascuno di questi, nell’ecologia ruminale, svolge un ruolo preciso, fondamentale e complesso che sta alla base dei processi digestivi. I batteri e gli archea, nonostante siano entrambi organismi monocellulari e procarioti, sono differenti ed appartengono a due domini distinti. Morfologicamente gli archea si distinguono dai batteri perché la loro membrana cellulare è costituita da eteri del glicerolo o del glicerolo fosfato, con catene isopreniche (alifatiche ramificate), mentre quelle batteriche sono costituite da esteri di fosfoglicerolo, con acidi grassi saturi o insaturi. Inoltre, nelle membrane degli archea non è presente l’acido muramico principale costituente del peptidoglicano, componente esclusivo della parete cellulare batterica, alla quale conferisce rigidità. Le principali differenze, però, si trovano a livello della trascrizione e traduzione del patrimonio genetico. Da questo punto di vista gli archea sono più simili agli eucarioti che non ai batteri stessi. Questi microrganismi, infatti, hanno tratti di r-RNA molto simili agli eucarioti e le loro polimerasi sono molto più complesse di quelle presenti nelle cellule batteriche e, a livello del DNA, presentano degli introni, ovvero delle sequenze di basi azotate non codificanti, come negli organismi più evoluti. Da un punto di vista strettamente metabolico, i batteri si distinguono dagli archea perché non producono metano mentre questa via metabolica è propria degli archea. Durante la degradazione anaerobica della componente fibrosa viene prodotta una grande quantità di H2, che a sua volta è trasformato dagli archea in metano (CH4) come una sorta di difesa, poiché il suo accumulo è nocivo per il microbiota. Questo viene poi eliminato mediante successiva eruttazione dell’animale. I metanogeni appartengono principalmente al phylum Euryarchaeota, all’interno del dominio Archaea. Gli Euryarchaeota sono attualmente suddivisi in 4 classi (Methanobacteria, Methanococci, Methanomicrobia e Methanopyri) e 7 ordini (Methanobacteriales, Methanococcales, Methanomicrobiales, Methanocellales, Methanosarcinales, Methanopyrales e Methanomassiliicoccales). I rapporti fra i microrganismi ruminali sono estremamente complessi e i vari gruppi microbici hanno tra loro relazioni di mutualismo o antagonismo. Inoltre, sono fortemente influenzati dalla dieta con particolare riferimento alla quantità e qualità della componente fibrosa. Molte delle attività dei microrganismi ruminali sono sinergiche, ovvero l’attività di alcuni di essi dipende dai metaboliti di altri. Un esempio è proprio la metanogenesi, che è operata dagli archea che riciclano l’idrogeno (H2) prodotto durante l’acetogenesi condotta dai cellulosolitici.
Principali vie biochimiche della metanogenesi nei ruminanti
I ruminanti vengono nutriti con un’ampia gamma di materiali di origine vegetale, tra cui erbe e legumi (alimentati freschi al pascolo o conservati mediante insilato o essiccamento), cereali (mais, orzo, avena, frumento), colture foraggere, scarti delle attività orticole (vinaccia, paglia di riso) o combinazioni di quanto sopra. Una volta che le materie prime per l’alimentazione zootecnica entrano nel rumine, vengono colonizzate principalmente dai batteri del rumine, che secernono enzimi che attaccano e distruggono i vari componenti della pianta. I polisaccaridi strutturali (cellulosa, emicellulose, pectina) vengono depolimerizzati in oligosaccaridi e zuccheri semplici, che vengono poi convertiti attraverso diversi schemi di fermentazione in acidi grassi volatili (VFAs), principalmente acetato, propionato e butirrato, e quantità minori di formiato, valerato, succinato, lattato e VFAs a catena ramificata, anidride carbonica (CO2) e idrogeno (H2). Anche i polisaccaridi non strutturali (amido, fruttani) e gli zuccheri solubili sono rapidamente fermentati a VFAs. I composti metilati, come le metilammine, i metilsolfuri e l’etanolo, possono anche essere rilasciati nel rumine dalla degradazione di lipidi vegetali, amminoacidi solforati o pectine, rispettivamente. A differenza di altri ambienti anaerobici ricchi di carbonio, i VFAs prodotti non vengono ulteriormente ossidati tramite reazioni microbiche sintrofiche nel rumine, poiché gli organismi che degradano i VFAs in composti più piccoli e H2 crescono e il tasso di ricambio del materiale attraverso il rumine è troppo alto per consentirne l’insediamento. Invece, i VFAs vengono assorbiti attraverso l’epitelio ruminale e vengono metabolizzati dal ruminante per l’energia e la crescita.
Una piccola gamma di questi prodotti finali di fermentazione può servire come fonte di energia per la crescita dei metanogeni, inclusi H2, formiato, acetato, alcoli a catena corta, metilammine e metilsolfuri. Il carbonio che funge da accettore di elettroni terminale nella formazione di CH4 può provenire da formiato, CO2, metanolo, composti metilici o dal gruppo carbonile dell’acetato. Poiché questi substrati sono prodotti finali della fermentazione di altri organismi che degradano composti organici più complessi, i metanogeni spesso formano associazioni stabili con questi organismi per facilitare i trasferimenti del substrato.
Esistono quattro modalità di formazione di CH4 riconosciute nei metanogeni (Fig.1):
- la via idrogenotrofica (consumo di H2) in cui l’idrogeno viene utilizzato per ridurre CO2 a CH4 in 7 passaggi enzimatici;
- la via metilotrofica H2-dipendente (consumo di composti metilici) dove H2 viene utilizzato per ridurre i composti metilici a CH4;
- la via metilotrofica H2-indipendente in cui i composti metilici possono essere, tramite una reazione di disproporzione, trasformati in CH4 e CO2;
- la via acetoclastica (scissione dell’acetato) in cui l’acetato viene metabolizzato per produrre CH4 e CO2.
Fig.1- Percorsi di metanogenesi nei metanogeni. MF, (metanofurano); Formyl-MF (formilmetanofurano), H4MPT, tetraidrometanopterina; F420, 8-idrossi-f-deazaflavina; CoM, coenzima M (2-sulfaniletansolfonato), CoB, coenzima B (7-mercaptoeptanoil-treoninfosfato).
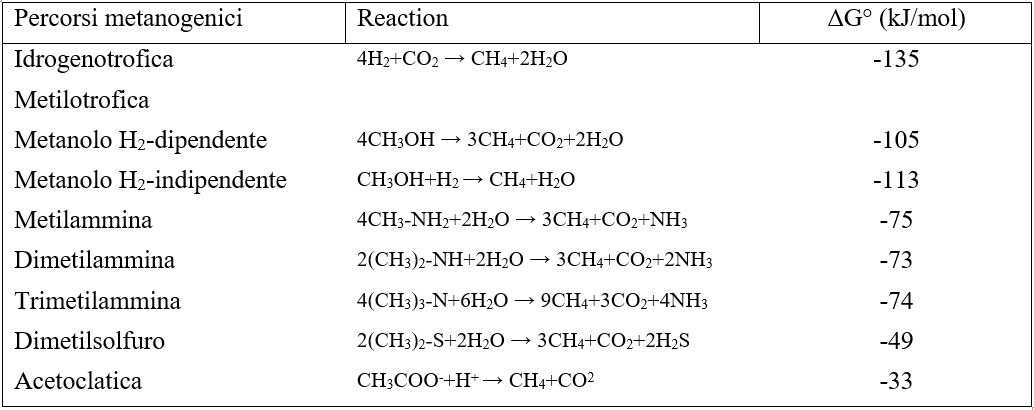
Ciascuno di questi percorsi differisce per la quantità di energia libera disponibile (Tabella 1).
Tabella 1- Energie libere di reazione associate alla metanogenesi ruminale e vie di utilizzo dell’idrogeno concorrenti.
Analisi quantitative indicano che l’H2 è la principale fonte di energia per la metanogenesi, sebbene il formiato possa contribuire fino al 18% del CH4 formato nel rumine. Anche le metilammine ed il metanolo possono essere usati da metanogeni metilotrofici dell’ordine Methanosarcinales, da Methanosphaera spp., dall’ordine Methanobacteriales e anche da membri del 7° ordine di metanogeni, i Methanomassiliicoccales.
Il contributo che i composti metilici danno alla metanogenesi ruminale non è stato misurato direttamente, ma è stato osservato che il metanolo infuso nel rumine delle pecore è stato rapidamente e completamente convertito in CH4, indicando che la disponibilità del composto metilico limita la metilotrofia nel rumine.
Le stime dell’abbondanza di metanogeni in grado di ridurre i gruppi metilici derivati dal metanolo indicano che circa il 22% dei metanogeni ruminali rilevati in un censimento globale potrebbero essere metilotrofi. Mentre l’acetato è presente ad alta concentrazione nel rumine (~ 60 mM), si ritiene che la formazione di CH4 dall’acetato sia trascurabile, poiché i metanogeni acetoclastici hanno tempi di raddoppiamento di 1-2 giorni e non sono in grado di crescere abbastanza velocemente da non essere lavati via dal rumine dove il tempo di ritenzione del liquido è solitamente inferiore a 1-2 giorni. Nel censimento globale del rumine, i metanogeni capaci di formare metano da acetato sono estremamente rari, rappresentando meno dello 0,015% di tutti gli Archaea. Tuttavia, i metanogeni che utilizzano acetato, come Methanosarcina, sono stati isolati dal rumine, ma di solito quando sono presenti substrati alternativi come metanolo o metilammine. I Methanosarcina spp. sono noti per crescere più rapidamente su metanolo o metilammine che su H2, e presumibilmente sostengono le loro popolazioni nel rumine crescendo più rapidamente quando questi substrati sono disponibili. I Methanosarcina compaiono in numero significativo anche quando il tasso di turnover del rumine è stato drasticamente rallentato, consentendo loro probabilmente di crescere principalmente utilizzando l’acetato.
Sebbene l’idrogeno sia la principale fonte di energia per la metanogenesi nel rumine, altri microrganismi idrogenotrofi possono competere per H2 in determinate condizioni ruminali. Gli omoacetogeni, che utilizzano H2 per ridurre la CO2 in acetato attraverso una via di acetogenesi, sono stati isolati dal contenuto del rumine e sono stati rilevati a basse concentrazioni in indagini molecolari. Tuttavia, gli omoacetogeni hanno soglie di H2 di ~350-700 nM, mentre i metanogeni idrogenotrofi hanno soglie molto più basse, tra 20 e 75 nM. La concentrazione di H2 disciolta nel rumine varia ampiamente a seconda della dieta, del tempo di alimentazione e dei tassi di turnover del rumine, ma di solito è compresa tra 400 e 3400 nM. Tipicamente, queste concentrazioni di H2 sono al di sopra della soglia richiesta per i metanogeni ma spesso inferiori alla soglia necessaria agli acetogeni. Inoltre, l’energia libera disponibile dalla metanogenesi idrogenotrofica (-135 kJ/mol) è maggiore di quella dalla acetogenesi riduttiva (-71,6 kJ/mol), assicurando che il metano sia la nicchia che utilizza H2 nel rumine. Altri potenziali competitor per l’idrogeno sono i riduttori di solfato e nitrato. Tuttavia, le concentrazioni di solfato e nitrato di solito sono troppo basse perché questi processi siano significativi nel rumine.
Coltivazione di Archaea metanogenici dal rumine
I metanogeni sono anaerobi stretti con requisiti di crescita molto specifici. I tentativi iniziali di coltivarli per consentirne l’identificazione furono ostacolati dalle difficoltà di raggiungere e mantenere l’anaerobiosi a un potenziale redox sufficientemente basso (-340 mV) per consentire ai metanogeni di crescere in vitro. Fu solo nel 1940, quando fu sviluppato un metodo anaerobico di agitazione dell’agar, che le colonie di metanogeni poterono essere ottenute in coltura. Robert Hungate perfezionò la preparazione di terreni pre-ridotti e sviluppò metodi per l’esclusione dell’ossigeno nella preparazione e sterilizzazione di un supporto privo di ossigeno, nonché metodi per l’inoculazione asettica e il trasferimento di microbi anaerobici in mezzi dove era possibile mantenere un potenziale di ossidazione/riduzione inferiore a -330 mV. Ciò comportò l’uso di un tampone di anidride carbonica-bicarbonato-carbonato per mantenere un pH vicino alla neutralità, l’uso di cisteina e l’impiego della resazurina come indicatore di ossidazione/riduzione. Rimuovere in modo asettico un tappo da una provetta di coltura in cui un metanogeno aveva generato una pressione negativa richiedeva un’abilità eccezionale da parte dell’operatore per prevenire la contaminazione o l’ingresso di ossigeno nella provetta. Le richieste di perfezione nell’uso di queste tecniche erano così elevate che solo pochi metanogeni erano stati isolati in coltura pura prima del 1974.
Lo sviluppo di procedure in cui i metanogeni potevano essere coltivati in un’atmosfera pressurizzata è stato un importante progresso nell’isolamento e nella coltura di questi organismi con una quasi totale eliminazione dei rischi di contaminazione, perdita del potenziale riducente e della purezza della cultura. Il primo metanogeno ruminale isolato, purificato e descritto in dettaglio è stato il Methanobacterium ruminantium. La designazione del genere in seguito è cambiata in Methanobrevibacter e l’attuale ceppo tipo di specie Methanobrevibacter ruminantium M1T(DSM 1093) è stato reisolato dal contenuto del rumine bovino perché il ceppo originale non è sopravvissuto in deposito. La sopravvivenza delle colture di metanogeno purificate e il loro mantenimento a lungo termine nelle raccolte di colture è un problema ancora oggi affrontato nei laboratori di microbiologia ruminale di tutto il mondo. Da questi primi esperimenti di coltivazione, un certo numero di metanogeni è stato isolato e, in alcune circostanze, caratterizzato dal contenuto ruminale di bovini e ovini in varie località del mondo, riflettendo spesso l’importanza locale di particolari specie di ruminanti.
L’uso di tecniche molecolari per l’identificazione e quantificazione dei metanogeni del rumine
I primi studi basati sulla coltivazione si basavano fortemente sulla determinazione di un piccolo numero di caratteristiche fenotipiche per identificare i metanogeni, e fu solo con l’emergere di approcci di biologia molecolare e l’analisi del DNA che i metanogeni poterono essere identificati inequivocabilmente attraverso tecniche di ibridazione e sequenziamento.
Le tecnologie NGS hanno indubbiamente un ruolo importante da svolgere nella futura caratterizzazione delle comunità di metanogeni ruminali. Con il termine Next Generation Sequencing (NGS), o sequenziamento in parallelo, si indicano una serie di tecnologie che permettono di sequenziare grandi genomi in un tempo ristretto. Si ottengono quindi informazioni sul DNA di organismi, animali e piante, fondamentali negli studi di genetica medica, genetica molecolare, genetica di popolazione, e genetica e genomica della conservazione. Ad oggi per il NGS ci sono diverse tecniche che si basano su principi differenti.
- Illumina: fluorescent sequencing, short reads
- Life Technologies: pH sequencing, si basa sulla variazione del pH che avviene quando viene incorporato un nucleotide
- Roche Parallel Pyrosequencing questo metodo è stato dismesso a partire dal 2015 perché era meno efficiente dei metodi basati sulla fluorescenza.
- Genereader NGS system (QIAGEN): sequenziamento basato su fluorescenza
- Pacific Bioscences SMRT: sequenziamento a singola molecola (single molecule, real time)
- Oxford Nanopore: sequenziamento a singola molecola basato sull’utilizzo di pori
- 10X Genomics: short reads (solitamente sequenziale su piattaforma Illumina) che appartengono fisicamente alla stessa molecola di DNA (linked reads)
Le tecnologie NGS stanno già rendendo routine il sequenziamento del genoma delle specie di metanogeni coltivate e stanno amplificando la nostra capacità di catalogare e analizzare i geni marcatori dei metanogeni. Stanno inoltre offrendo nuovi potenti strumenti per recuperare le sequenze metagenomiche dei metanogeni a profondità sempre maggiori, consentendo così l’accesso alle informazioni genomiche direttamente dai campioni di rumine tramite riassemblaggi del metagenoma.
Per la prima volta, questo approccio ha fornito dati quantitativi sugli Archaea totali nei campioni di rumine ed è stato utilizzato per caratterizzare i tipi di metanogeni presenti in una varietà di animali.