Già da un po’ di tempo si sente parlare di Fenomica, un altro termine della famiglia ‘omica’ che però suscita particolare interesse perché non riguarda direttamente la biologia molecolare ma il suo risultato finale: il fenotipo.
Introduzione
Negli ultimi 10 anni, anche in zootecnia, è diventato sempre più frequente sentir parlare di scienze ‘omiche’, intendendo con questo tutte quelle discipline che si occupano di studiare ed approfondire un determinato sistema biologico attraverso l’utilizzo di una grande mole di informazioni, generate spesso in tempi molto ridotti. Un esempio ben noto a tutti è la Genomica, che a partire da una serie di informazioni raccolte sul genoma degli animali da reddito, è in grado di fornire strumenti estremamente efficienti per migliorare l’accuratezza delle valutazioni genetiche. I risultati ottenuti sono ben evidenti agli occhi di tutti (Wiggans & Carrillo, 2022). Alla Genomica, se ne sono aggiunti altri come la proteomica, la metagenomica, la metabolomica o la trascrittomica. Tutte queste metodologie si stanno rivelando estremamente interessanti ma tutte hanno anche mostrato alcuni limiti. Ad esempio, nel caso della Genomica, i vantaggi sono stati veramente notevoli ma questi si sono ridotti di intensità, nonostante si stiano utilizzando anche le sequenze complete.
Uno dei motivi è che tutti questi approcci, per diventare applicativi e sfruttare appieno le loro potenzialità, hanno bisogno di dati, ma non di dati qualsiasi: hanno bisogno di dati “fenomenali”, hanno bisogno della nuova arrivata la Fenomica.
Definiamo la Fenomica
La definizione per così dire “ufficiale” di cosa si intenda per Fenomica risale al 2010 ed è stata data da Houle et al (2010) che la descrissero come “l’acquisizione su larga scala di dati fenotipici multi-dimensionali”. Gli stessi autori sottolinearono anche come la sua nascita fosse stata favorita dal sempre maggiore utilizzo di sensori elettronici che raccoglievano in tempo reale e continuo un’immensa mole di informazioni associate alla possibilità di condividerle, sempre in tempo reale, via internet.
Il primo riferimento indiretto alla Fenomica in zootecnia è stato fatto nel 2011 dal Dr. Mike Coffey durante un meeting di ICAR (presentazione disponibile a questo link). In quell’occasione il Dr. Coffey presentò il suo nuovo motto: “In the age of the genotype, phenotype is king” (“Nell’era dei genotipi, il re è il fenotipo”). Con questa semplice affermazione il Dr. Coffey aveva centrato l’obiettivo: inutile avere strumenti super accurati se non si hanno i dati da cui partire per poterli sfruttare, è come avere un set completo di posate quando da mangiare c’è solo un panino.
C’è anche un altro aspetto che rende però la Fenomica diversa dalle sue sorelle di suffisso: se ad esempio il genoma, seppur nella sua complessità, è ben definito (i cromosomi, gli snp, le basi azotate), lo stesso non si può dire per il fenotipo, che può assumere definizioni diverse a seconda di quanto, come, quando e dove sia stato misurato, e quindi diventa estremamente più eterogeneo. Si pensi ad esempio ai fenotipi grasso e proteine del latte, il loro rapporto ne genera un altro che può essere usato per valutare il bilancio energetico negativo. Oppure si pensi ai dati che cambiano nel tempo, come ad esempio l’evoluzione del microbiota.
Esempi di Fenomica
Quando si parla di Fenomica, in molti pensano a qualcosa di estremamente complicato o diverso rispetto ai fenotipi che abbiamo sempre utilizzato. In realtà non è così, è solo un modo diverso e più preciso di guardare le cose che abbiamo sempre osservato. Per usare un altro esempio, è come la differenza tra un moderno telefono cellulare ed un vecchio apparecchio di telefonia fissa: entrambi fanno la cosa basica – telefonare – ma il cellulare ci permette di fare molte altre cose utili (non proprio tutte).
Ecco quindi che oggi possiamo “guardare” un animale e raccogliere, attraverso adeguati sensori, molte più informazioni rispetto a prima. Questa nuova visione di insieme è ottimamente sintetizzata nella figura 1, tratta da Halachmi et al (2019).
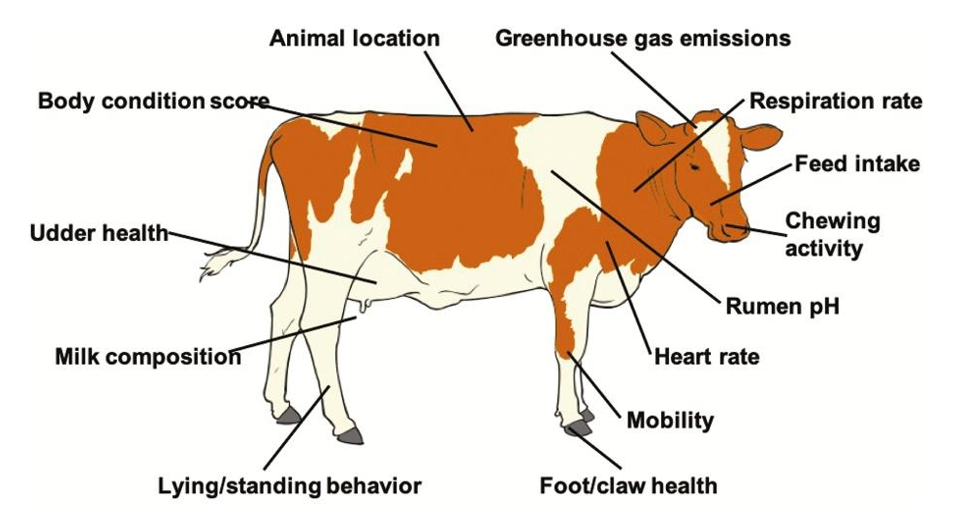
Figura 1. Fenotipi innovativi che possono essere raccolti a livello di singolo individuo da sensori specifici (Halachmi eta l, 2019).
Un esempio di come qualcosa di “tradizionale” possa essere rivisitato dalla Fenomica è la valutazione morfologica o la condizione corporale. Questi fenotipi sono estremamente importanti nell’ottica del benessere animale ma spesso forniscono informazioni limitate perché raccolti una sola volta nella vita dell’animale. Oggi questo aspetto può essere superato attraverso l’utilizzo di immagini raccolte ripetutamente nell’arco della giornata, delle settimane e dei mesi attraverso sensori collocati negli impianti di mungitura, nei robot o nelle zone di alimentazione/passaggio degli animali (Nye et al 2020).
La Fenomica ha però portato innumerevoli vantaggi in tutto ciò che vedevamo anche prima ma che non eravamo in grado di misurare. Anche aspetti “fuori dagli schemi”, come ad esempio il comportamento degli animali, tra loro e verso l’uomo. Oggi i caratteri del comportamento possono essere misurati e quantificati grazie alle immagini od ai sensori di movimento (Chen et al, 2020; Fernandes et al, 2020) e questo permette, ad esempio, la stima di una loro eventuale componente genetica.
Altri esempi di Fenomica sono ancor più attuali e riguardano tutti quegli aspetti legati direttamente od indirettamente alle emissioni di gas da parte dei ruminanti (Lu et al, 2017). Ecco quindi che la misurazione delle emissioni individuali o di gruppo e l’efficienza alimentare – sempre individuale o di gruppo – rappresentano nuovi fenotipi da raccogliere e valutare, sempre però nell’ottica di avere misurazioni nel tempo e su stadi fisiologici diversi.
Considerazioni Finali
Come spesso succede, tutte le novità portano entusiasmo ed a volte si viene travolti dall’entusiasmo. La Fenomica sta producendo davvero una mole enorme di informazioni e si potrebbe correre il rischio di diventare degli accumulatori seriali. In questo senso nuovi approcci metodologici e di archiviazione sono necessari per gestire e analizzare questa mole di dati. Ecco quindi che l’Intelligenza Artificiale ritorna ad essere al centro dell’attenzione, come lo strumento più efficace ed efficiente per apprendere da questi dati e per poterli trasformare in applicazioni di campo. Allo stesso tempo l’archiviazione “fisica” sta lasciando il posto, o viene sempre più affiancata, da un’archiviazione “virtuale”, con spazi su cloud dedicati.
Un ultimo aspetto riguarda la proprietà dei dati. Spesso le informazioni raccolte dai sensori posti sui nostri animali sono di proprietà della ditta che ha creato e/o commercializza il determinato sensore, e questo pone dei limiti ad una eventuale condivisione dell’informazione o per lo meno alla sua associazione con altre informazioni. Un aspetto da non sottovalutare per evitare di “fare i conti senza l’oste”.
Bibliografia Consultata
Chen C, Zhu W, Steibel J, Siegford J, Wurtz K, Han J, et al. Recognition of aggressive episodes of pigs based on convolutional neural network and long short-term memory. Comput Electron Agric. 2020;169:105166.
Fernandes AFA, Dórea JRR, de Rosa GJ. Image analysis and computer vision applications in animal sciences: an overview. Front Vet Sci. 2020;7:551269.
Halachmi, I., M. Guarino, J. Bewley, and M. Pastell. 2019. Smart animal agriculture: application of real-time sensors to improve animal well-being and production. Annu. Rev. Anim. Biosci. 7:403–425. doi:10.1146/annurev-animal-020518-114851.
Houle D, Govindaraju DR, Omholt S. Phenomics: the next challenge. Nat Rev Genet. 2010;11:855–66.
Lu D, Jiao S, Tiezzi F, Knauer M, Huang Y, Gray KA, et al. The relationship between different measures of feed efficiency and feeding behavior traits in Duroc pigs. J Anim Sci. 2017;95:3370–80.
Negussie E, Lehtinen J, Mäntysaari P, Bayat AR, Liinamo AE, Mäntysaari EA, et al. Non-invasive individual methane measurement in dairy cows. Animal. 2017;11:890–9.
Wiggans, G. R., & Carrillo, J. A. (2022). Genomic selection in United States dairy cattle. Frontiers in Genetics, 13, 994466.
Nye J, Zingaretti LM, Pérez-Enciso M. Estimating conformational traits in dairy cattle with DeepAPS: a two-step deep learning automated phenotyping and segmentation approach. Front Genet. 2020;11:513.








Scrivi un commento
Devi accedere, per commentare.