Nelle ultime settimane si è parlato molto di Intelligenza Artificiale (IA), questo soprattutto in conseguenza delle controversie nate attorno all’uso di un software in grado di simulare ed elaborare le conversazioni umane. In realtà le applicazioni di Intelligenza Artificiale sono molteplici, ed anche nel campo del miglioramento genetico rappresentano un’interessante alternativa ed un campo di ricerca innovativo.
Introduzione
Grazie alle innovazioni raggiunte nel campo dell’hardware (i.e. delle componenti fisiche dei computer), delle infrastrutture (e.g. cloud computing), degli algoritmi di calcolo e della generazione di grandi moli di dati (e.g. sequenze genomiche, real-time data), l’utilizzo di processi di Intelligenza Artificiale (AI) come il deep learning (DL) sta incontrando enorme successo nel campo della predizione. Questo avviene negli ambiti più disparati, da quello medico con applicazioni di medicina di precisione (Johnson, K. B. et al. 2021) a quello agricolo e zootecnico con la oramai ben nota agricoltura e zootecnia di precisione (Mahmud et al, 2021, De Alwis, S. et al., 2022).
Il successo di questo approccio risiede anche nel fatto che il DL è favorito dalla sua versatile capacità di gestire dati cosiddetti “non strutturati” e particolarmente “complessi”. Immaginiamo di osservare un numero qualsiasi, tutti lo riconosceremo allo stesso modo, con il suo valore. Ma se tutti osserviamo la stessa fotografia ognuno di noi potrebbe avere una reazione diversa perché l’immagine non ha una struttura univoca o con un’unica interpretazione. Quello che avviene guardando una fotografia avviene anche con le caratteristiche di interesse zootecnico, dove ciò che osserviamo, il fenotipo, è il risultato della combinazione e relativa interazione di diverse fonti di variazioni, come quelle ambientali e genetiche. Ognuna avrà il suo contributo, che varierà nel tempo e dipenderà da diverse interazioni (e.g. interazione genotipo x ambiente). Non a caso negli ultimi anni l’introduzione delle informazioni legate al genoma degli animali da reddito ha sicuramente migliorato l’accuratezza delle nostre predizioni, e cioè dei valori genetici dei singoli riproduttori, a scapito però della complessità del sistema di calcolo.
Cosa intendiamo per Intelligenza Artificiale e Deep Learning
Nonostante se ne parli davvero tanto, non tutti hanno ben chiaro cosa si intenda per Intelligenza Artificiale (AI) e Deep Learning. Non è questa certo la sede per raccontare come e dove nasce l’AI, però una definizione che possiamo considerare autorevole è quella dell’Organizzazione per la Cooperazione e lo Sviluppo Economico (OCSE) che lo definisce come “un sistema basato su macchine progettato per funzionare con diversi livelli di autonomia e che può, per obiettivi espliciti o impliciti, generare output come previsioni, raccomandazioni o decisioni che influenzano ambienti fisici o virtuali”. Nell’ambito di questo sistema esistono poi diversi processi che possono essere utilizzati per rendere “applicabile” l’AI. Il Deep Learning è uno di questi, e deve la sua notorietà al fatto di “operare” come un sistema a più livelli, definiti reti neurali, che ricorda molto da vicino il funzionamento del cervello umano: una serie di stimoli esterni, dei recettori che captano questi stimoli, un sistema che si attiva interpretando e “pesando” gli stimoli in sequenza, ed una risposta finale mediata da questi stimoli a cascata.
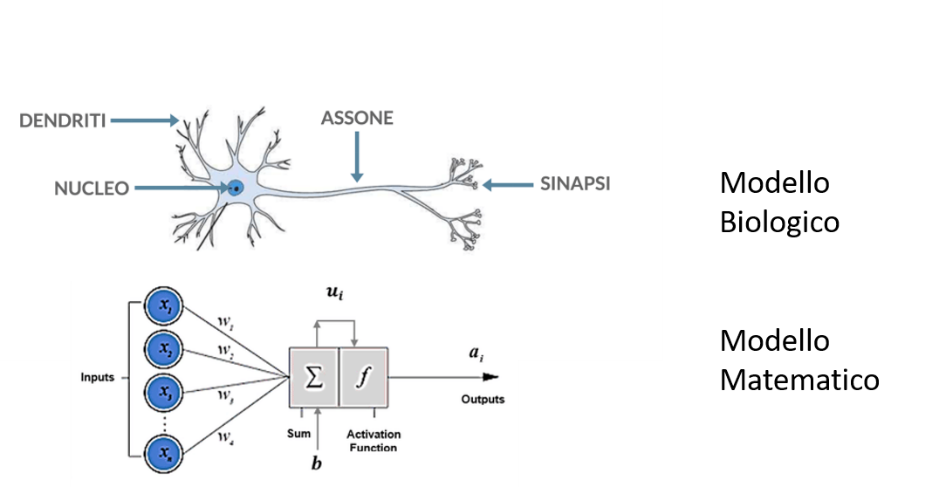
Nella figura 1 sono rappresentati uno accanto all’altro un neurone ed una rete neurale. Il neurone è composto da alcune parti – dendriti, soma e assone – che lo connettono agli altri neuroni e che ricevono, elaborano e trasmettono un certo input. La rete neurale ha invece dei nodi di ingresso (paragonabili ai dendriti) e di uscita (similmente agli assoni), oltre che delle funzioni di elaborazione degli input ricevuti.
Ad esempio, tornando al mondo animale, gli input della nostra rete neurale potrebbero essere rappresentati dai marcatori molecolari oppure dallo stadio di lattazione, o da entrambe le cose contemporaneamente (e quindi anche dalla loro interazione).
Figura 1. Confronto tra la struttura di un Neurone (Modello Biologico) ed una rete Neurale (Modello Matematico). Gli inputs di una rete neurale potrebbero, ad esempio, essere dei marcatori molecolari oppure lo stadio di lattazione di una bovina.
Primi risultati
Diversi ricercatori hanno cominciato ad applicare il DL al mondo animale, ed in particolare alla stima dei valori genetici e genomici. Una prima applicazione su dati reali di suini è stata quella di Waldmann (2018) che, utilizzando dati di 3534 suini, ognuno genotipizzato con 50276 SNPs e con un fenotipo di accrescimento con un’ereditabilità pari a 0.38, ha stimato il loro valore genomico con delle reti neurali confrontandoli con metodi più tradizionali, come il GBLUP, approccio attualmente utilizzato in diverse parti del mondo e su diverse specie da reddito. I risultati sono stati estremamente interessanti e le stime ottenute attraverso il DL hanno presentato un errore minore, una minore tendenza alla sovrastima ed anche la capacità di fornire stime dell’importanza di ciascun marcatore. Questo ultimo aspetto è particolarmente importante perché spesso uno dei limiti dei metodi di DL è proprio la scarsa interpretabilità dei risultati intermedi.
Un altro approccio molto interessante, anch’esso ottenuto con dati di genotipizzazione reali e disponibili attraverso un dataset pubblico (genotipi di 3 diverse razze bovine, van den Berg et al., 2015,), è stato quello di Nazzicari e Biscarini (2022). L’idea innovativa del loro lavoro è stata quella di utilizzare una classe specifica di DL, un cosiddetto deep convolutional neural network, normalmente utilizzato per analizzare la struttura di video ed immagini, per ridimensionare la matrice di parentela ed utilizzarla per la stima di valori genomici di una serie di fenotipi quantitativi e qualitativi. I risultati ottenuti sono stati quindi confrontati, anche questa volta, con il metodo GBLUP, che rappresenta il gold standard dei metodi di calcolo dei valori genomici e che utilizza una matrice di parentela genomica calcolata a partire dai marcatori molecolari disponibili. In questo caso i risultati sono stati contrastanti dipendendo dal carattere analizzato (quantitativo o qualitativo) e dalla misura utilizzata per valutare la bontà del metodo (errore medio o correlazione).
Infine, Hamadani et al (2022) hanno utilizzato ben 13 diversi approcci di AI per stimare i valori genetici del peso corporale di pecore di razza Corriedale, i cui dati erano stati raccolti dal 1969 al 2021. In questo caso l’accuratezza delle reti neurali variava da un minimo di 0.621 ad un massimo di 0.852.
Conclusioni
Il progresso genetico degli animali da reddito dipende fortemente dall’accuratezza con la quale stimiamo i nostri strumenti selettivi, cioè gli indici genetici. Negli ultimi 10 anni, con l’introduzione della genomica, abbiamo assistito a dei miglioramenti formidabili. Tuttavia, altre sfide sono già pronte, soprattutto in conseguenza della nuova tendenza di fenotipizzazioni automatizzate, che permettono di raccogliere dati in maniera continua e dettagliata.
In quest’ottica l’utilizzo di metodi di Intelligenza Artificiale, come ad esempio il deep learning, rappresentano uno strumento in più. La loro applicazione per la stima dei valori genetici è già iniziata ma diversi sono gli aspetti da considerare prima che diventino lo strumento ideale. Tra questi la necessità di una grande mole di dati, al fine di evitare quello che rappresenta il problema principale di questi approcci: il cosidetto overfitting e cioè il sembrare molto più bello di quello che realmente è. Inoltre ci sono anche problemi di interpretazione e spesso non si è in grado di spiegare bene il perché di un certo risultato. Per chi lavora “in campo”, capire cosa sta succedendo e perché è fondamentale. Però la tecnica è eccezionale ed è molto più utile ed efficace che scrivere in una semplice chat aspettandosi delle risposte.
Bibliografia
De Alwis, S. et al. A survey on smart farming data, applications and techniques. Comput. Ind. 138, 103624 (2022).
Hamadani, A., Ganai, N.A., Mudasir, S. et al. Comparison of artificial intelligence algorithms and their ranking for the prediction of genetic merit in sheep. Sci Rep 12, 18726 (2022). https://doi.org/10.1038/s41598-022-23499-w
Johnson, K. B. et al. Precision medicine, AI, and the future of personalized health care. Clin. Transl. Sci. 14, 86–93 (2021).
Mahmud, M. S., Zahid, A., Das, A. K., Muzammil, M. & Khan, M. U. A systematic literature review on deep learning applications for precision cattle farming. Comput. Electron. Agric. 187, 106313 (2021).
Nazzicari, N. and Biscarini, F., 2022. Stacked kinship CNN vs. GBLUP for genomic predictions of additive and complex continuous phenotypes. Scientific Reports, 12(1), p.19889.
van den Berg, S. et al. Across population genomic prediction scenarios in which Bayesian variable selection outperforms gblup. BMC Genet. 16, 1–12 (2015).
Waldmann, P. Approximate Bayesian neural networks in genomic prediction. Genet. Sel. Evol. 2018, 50, 70








Scrivi un commento
Devi accedere, per commentare.