Negli ultimi anni molti di noi avranno sicuramente sentito parlare di Epigenetica, cioè di quella scienza che cerca di capire come fattori non genetici ma ereditabili modifichino l’espressione di un gene, e quindi il fenotipo, senza alterare però il genotipo. Un pò come fanno gli accenti in una parola. Dopo molte promesse, l’avvento di nuove tecnologie potrebbe rendere questa scienza applicabile su grande scala anche alla zootecnia.
Sicuramente non ci facciamo mai caso ma ci sono molte parole, dette omografe, che pur essendo esattamente identiche perché composte dalla stessa sequenza di lettere, in realtà contengono una piccola differenza, l’accento, che ne cambia completamente il significato. Pensiamo a Prìncipi e Princìpi: nel primo caso siamo di fronte al plurale di “principe” mentre nel secondo al plurale di “principio”. Due cose ben diverse.
Nel genoma succede qualcosa di simile. A causa di alcune molecole (che chiameremo gli accenti del genoma) le stesse sequenze possono essere lette in maniera diversa, e quindi la loro espressione – che non è altro che il fenotipo – risulta diversa.
Quante volte ci siamo domandati perché individui geneticamente identici o comunque molto simili possano avere fenotipi estremamente diversi. È ovvio che c’è l’effetto dell’ambiente ma c’è anche qualcosa che probabilmente sta sopra alla genetica ed è in grado di cambiare le cose senza cambiarle: in greco il prefisso “epi” significa proprio “sopra” e da qui nasce il termine Epigenetica.
In concreto, le modifiche epigenetiche avvengono grazie a una serie di molecole – gli accenti epigenetici – che, tramite specifiche reazioni, si attaccano o si staccano da specifiche porzioni del DNA, rendendole più o meno accessibili. Tali modifiche variano per tipo di gene, di cellula e inoltre nel tempo.
Conseguenza delle modifiche epigenetiche è il fatto che un gene sia o meno trascritto in RNA messaggero, e dunque tradotto o meno in una proteina. A seconda che tale proteina venga o non venga prodotta, essa potrà o meno esercitare la propria funzione e quindi modificare il fenotipo – ciò che vediamo.
Un tipo di accento: la metilazione del DNA
Come anticipato, esistono diverse forme di accenti o, volendo essere più rigorosi nei termini, di meccanismi epigenetici. Tra questi, la metilazione del DNA, che consiste nell’aggiunta di un gruppo chimico (metile, formula -CH3) in punti specifici del DNA, è uno dei più studiati. Questo anche grazie alle tecniche di sequenziamento utilizzate per identificarlo.
In genere la metilazione blocca l’espressione del gene e di fatto lo inattiva, per esempio impedendo ad apposite proteine di trascrivere il DNA (figura 1).
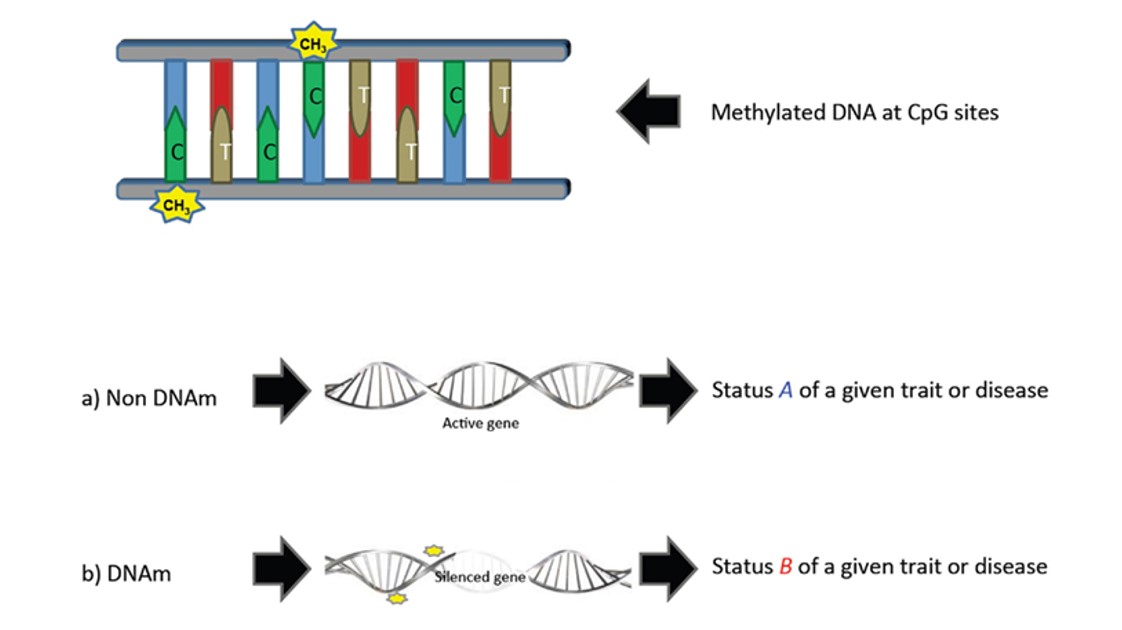
Figura 1. Esempio di Metilazione del DNA. Un gruppo metile si lega ad una base pirimidinica (citosina) e modifica l’espressione del carattere dallo stato A a quello B. Fonte: González-Recio, Oscar. “Epigenetics: a new challenge in the post-genomic era of livestock.” Frontiers in genetics 2 (2012): 106.
Già da alcuni anni l’epigenetica è finita al centro delle attenzioni di chi si occupa di miglioramento genetico. Non solo perché potrebbe aiutare a spiegare quella che in gergo viene chiamata come la “missing heritability” dei caratteri complessi, e cioè quella parte di variabilità genetica che non si riesce a spiegare neanche conoscendo tutto il genoma, ma anche perché i famosi accenti possono comparire o modificarsi nel corso della vita di un individuo in conseguenza di fattori ambientali più o meno invasivi (Petronis, 2010). Conoscerli vuol dire poter stimare meglio gli effetti genetici additivi, che ci servono per fare selezione.
Non solo, diverse ricerche hanno oramai dimostrato come gli effetti della metilazione si propagano verticalmente tra individui che in un certo punto della loro vita coabitano, come nel caso di una femmina in produzione, del suo feto e delle cellule germinali di quest’ultimo (figura 2).
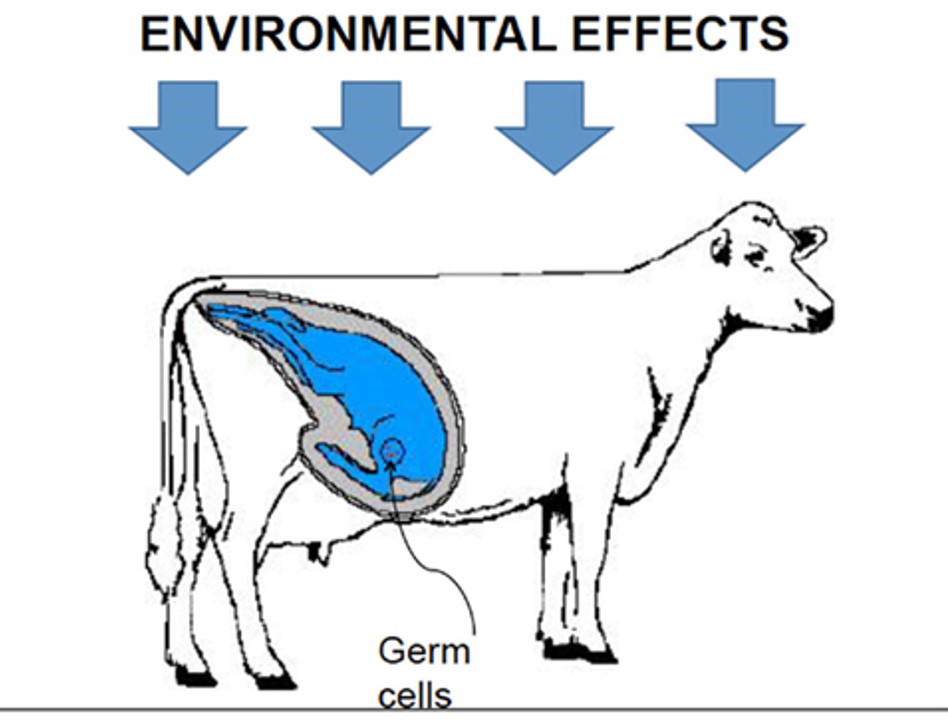
Figura 2. I fattori ambientali al tempo t possono influenzare i meccanismi di metilazione di tre generazioni. Fonte: González-Recio, Oscar. “Epigenetics: a new challenge in the post-genomic era of livestock.” Frontiers in genetics 2 (2012): 106.
Ecco quindi che quello che succede ad un individuo durante la sua vita può avere conseguenze nelle generazioni future.
Un recente studio condotto da Macciotta e colleghi utilizzando dati della Pezzata Rossa Italiana (Macciotta et al, 2023) ha dimostrato come le pronipoti di vacche che hanno partorito in estate od in autunno hanno avuto performance produttive inferiori a pronipoti di vacche che hanno invece partorito in primavera o inverno.
Lo studio è stato condotto utilizzando come fenotipi i valori genetici delle pronipoti ma ciononostante è un’indicazione molto interessante di un possibile effetto epigenetico transgenerazionale.
I limiti e le nuove possibilità
Nonostante le enormi potenzialità ed il forte interesse del mondo scientifico verso l’epigenetica, esistono una serie di problematiche che ne complicano l’utilizzo e ne rendono più complessa e lenta la sua eventuale applicazione in campo.
Un primo problema riguarda il modo in cui si esprime la metilazione, espressione che cambia nel tempo entro individuo e tra individui della stessa generazione. In questo senso l’ideale sarebbe l’utilizzo di dati ripetuti o, sempre per usare termini più scientifici, dati longitudinali.
Un altro aspetto riguarda la proporzione di varianza o variabilità fenotipica che l’epigenetica può spiegare e per quante generazioni.
C’è poi la questione legata al tipo di tessuto sul quale si vuole studiare l’effetto della metilazione, anche perché l’effetto è tessuto specifico. Se, ad esempio, l’interesse è legato al miglioramento genetico, allora l’ideale sarebbe studiare l’effetto della metilazione sulle cellule germinative e quindi su quel tipo di cellule che si differenzieranno in cellule riproduttive.
Ecco, tutti questi aspetti, di per sé relativamente risolvibili, dipendono però tutti da un punto fondamentale: essere in grado di identificare le regioni metilate attraverso la lettura del genoma.
I primi metodi in grado di identificare regioni del genoma metilate sono apparsi agli inizi degli anni ’90 grazie all’avvento del cosiddetto sequenziamento a bisulfito (Frommer et al., 1992), metodi questi superati dai metodi di sequenziamento di seconda generazione, molto più efficienti e con maggiore risoluzione (Laird, 2010). Con un problema però: i costi elevati e la necessità di sequenze complete.
Questo vorrebbe dire sequenziare il genoma completo di tanti individui, un approccio impraticabile per i costi, la dimensione dei dati prodotti ed i tempi di analisi.
Un recente studio condotto in Spagna da González-Recio ed il suo gruppo (González-Recio, 2023) ha però presentato dei risultati estremamente interessanti attraverso l’applicazione concomitante di due tecnologie di avanguardia su bovini da latte: la genotipizzazione attraverso il sequenziamento – nota come Genotype by Sequencing – ed un sequenziamento di terza generazione, il sequenziamento Nanopore.
Il sequenziamento Nanopore si basa sull’uso di nanopori inseriti in una membrana elettro-resistente, attraversata da una corrente ionica. Quando una molecola di DNA/RNA passa attraverso il poro, causa una specifica alterazione nel flusso di corrente che dipende dalle basi (di solito 3-meri) presenti nel poro.
Durante il passaggio della molecola di DNA/RNA attraverso il poro, vengono fatte numerose misurazioni, e l’alterazione di corrente viene convertita da un software di basecalling nella corrispettiva sequenza genomica (tratto da https://www.microbiologiaitalia.it/strumentazioni/sequenziamento-nanopore/).
In particolare, quest’ultima tecnologia sembra essere la chiave di volta di eventuali applicazioni su grande scala dell’epigenetica. Oltre infatti ad avere costi piuttosto contenuti ed a permettere la genotipizzazione che tutti oramai conosciamo, questa tecnologia permette di ottenere in contemporanea ed allo stesso costo anche le informazioni sulla metilazione di un elevato numero di campioni.
Queste informazioni, contrariamente a quanto avveniva prima, potrebbero così essere usate non solo per studi di associazione tra l’epigenoma e fenotipi di interesse – Epigenome Wide Association Studies – ma potrebbero anche essere inserite nei ben noti modelli misti utilizzati nella stima dei valori genetici con lo scopo di ottenere predizioni ancor più precise.
Considerazioni Finali
Durante gli ultimi anni abbiamo assistito a dei progressi tecnologici davvero impressionanti. Progressi che hanno consentito l’applicazione di metodologie eccezionali. La genomica ne è un esempio cristallino. La ricerca, come al solito, non si è fermata, e nonostante a volte si faccia prendere dall’euforia e perda di vista l’eventuale applicabilità, i risultati che si possono raggiungere sono davvero enormi.
La genetica e la genomica ci stanno spiegando molte cose ma altre rimangono ancora difficili da capire e cogliere. L’epigenetica si muove in questo mondo più ignoto, ma come tutti quelli che si avventurano in un mondo sconosciuto c’è bisogno di strumenti adeguati per poterlo fare.
Fino a ieri l’epigenetica era un pò considerata l’eterna promessa, bella ma un pò inconsistente. Oggi grazie ai miglioramenti tecnologici raggiunti, potrebbe diventare un ulteriore alleato nella comprensione dei tanti aspetti che determinano la comparsa e l’evoluzione di fenotipi di interesse.
Bibliografia consultata
- Frommer, M., McDonalD, L. E., Millar, D. S., Collis, C. M., Watt, F., Grigg, G. W., Molloy, P. L., and Paul, C. L. (1992). A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827–1831.
- González-Recio, Oscar. “Epigenetics: a new challenge in the post-genomic era of livestock.” Frontiers in genetics 2 (2012): 106.
- González-Recio, O., López-Catalina, A., Peiró-Pastor, R., Nieto-Valle, A., Castro, M., & Fernández, A. (2023). Evaluating the potential of (epi) genotype-by-low pass nanopore sequencing in dairy cattle: a study on direct genomic value and methylation analysis. Journal of Animal Science and Biotechnology, 14(1), 98.
- Laird, P. W. (2010). Principles and challenges of genome-wide DNA methylation analysis. Nat. Rev. 11, 191–203.
- Macciotta NPP, Dimauro C, Degano L, Vicario D, Cesarani A. A transgenerational study on the effect of great-granddam birth month on granddaughter EBV for production traits in Italian Simmental cattle. J Dairy Sci. 2023 Apr;106(4):2588-2597. doi: 10.3168/jds.2022-22455. Epub 2023 Mar 2. PMID: 36870840.
- Petronis, A. (2010). Epigenetics as a unifying principle in the aetology of complex traits and diseases. Nature 465, 721–727.







Scrivi un commento
Devi accedere, per commentare.