
Negli ultimi anni si parla con sempre maggiore frequenza di “microbiota”, cioè di quella comunità composta da miliardi di microorganismi che vive nel tratto gastrointestinale degli esseri viventi. Nei ruminanti è ancor più importante considerati i legami sia con l’efficienza alimentare che con altri caratteri produttivi e non solo.
È possibile quantificare l’effetto del “microbiota” sul fenotipo?
Introduzione
André Godin, un industriale francese nonché studioso di problemi sociali vissuto nel XIX secolo, diceva che “le parole sono i chiodi per attaccare le idee”. Se c’è una parola che negli ultimi anni di idee ne ha generate tante, e a volte le ha anche confuse, questa è sicuramente microbiota. In realtà il termine in sé è relativamente semplice, perché per microbiota si intende l’insieme di miliardi di microorganismi – batteri, protozoi e funghi – che vivono nel tratto gastrointestinale degli esseri viventi (figura 1).

Figura 1. Microbioma Umano (fonte: https://www.genome.gov/genetics-glossary/Microbiome) e Animale (Fonte:https://www.biomedcentral.com/collections/animalgut)
L’interesse crescente, direi quasi esponenziale, che c’è stato nei suoi confronti è legato agli effetti che il microbiota ha sulle performance degli individui, siano esse produttive, riproduttive o di benessere e resilienza (Hou et al, 2022; Matthews et al., 2019).
Negli animali da reddito l’interesse è stato ancora maggiore, soprattutto nel caso dei ruminanti i quali, come ben sappiamo, possiedono una popolazione microbica ad-hoc che gli permette di utilizzare alimenti composti principalmente da lignocellulòsa e di trasformarli in carne e latte poi utilizzati per alimentazione umana. Nel caso specifico dei ruminanti, l’interesse è ancora maggiore per via del rapporto tra attività della flora microbica ruminale ed emissioni di metano, rapporto alla base della nota campagna di demonizzazione in atto nei confronti di questa categoria di animali da reddito.
L’interesse per questa affascinante e poco conosciuta comunità è stato anche favorito dalla disponibilità di nuove tecniche di analisi come la cosiddetta metagenomica, che utilizza il sequenziamento e l’isolamento del DNA per identificare i diversi microorganismi presenti, e la loro frequenza, distribuzione e rapporto.
A partire dai risultati ottenuti attraverso queste tecniche è stato possibile cominciare a studiare e valutare i rapporti tra il microbiota, chi lo ospita (e.g. la vacca da latte) ed il risultato finale (e.g. efficienza alimentare, produzione di latte, salute).
Il concetto di Microbiabilità
Uno dei concetti più noti ed alla base del miglioramento genetico è l’ereditabilità, parametro che esprime e quantifica il rapporto tra la genetica (nella sua componente additiva) e la variazione osservata nel fenotipo. Così come le variazioni nel genoma di un animale influenzano il fenotipo, allo stesso modo le variazioni della comunità di microorganismi, ospiti del succitato animale, anch’esse hanno un effetto sul fenotipo.
È sulla base di queste osservazioni che nel 2016 Difford ed i suoi colleghi (Difford et al, 2016) hanno studiato e quantificato l’effetto del microbiota sui fenotipi, coniando il termine Microbiabilità. Questo è stato possibile applicando le stesse metodologie utilizzate nel miglioramento genetico classico o nell’attuale genomica, dove le relazioni genetiche/genomiche tra gli individui vengono utilizzate e sfruttate attraverso una cosiddetta matrice di parentela.
Questa famosa matrice di parentela è stata quindi sostituita da una cosiddetta microbial relationship matrix (che potremmo approssimativamente tradurre in matrice di parentela microbiale) che ha permesso di stimare la componente di variabilità dovuta al microbiota e quindi il suo rapporto con la variabilità totale o fenotipica, cioè la Microbiabilità (Ross et al., 2021; Difford et al, 2018).
Alcuni esempi dai bovini da latte, dagli ovini e dai suini
A partire dalla definizione di questo nuovo parametro genetico, molti studi sono stati condotti e si stanno ancora conducendo per capire l’importanza, il ruolo e l’eventuale applicazione delle informazioni provenienti dal microbiota.
Difford et al, nel loro lavoro del 2018, si sono focalizzati sugli aspetti legati alla produzione di metano nelle vacche da latte trovando che la variazione dovuta al microbiota ruminale – i.e. la Microbiabilità – era pari al 13%, contro il 21% di quella dovuta al genoma dell’ospite (ereditabilità). I loro risultati hanno anche dimostrato che, contrariamente a quanto inizialmente ipotizzato, il genoma dell’ospite non influenza il genoma del microbiota.
In un recente lavoro, sempre nelle vacche da latte, Boggio e colleghi (2023a) hanno affrontato gli effetti del genoma, del micriobioma e della loro interazione (figura 2) sull’ingestione di sostanza secca, sul valore energetico del latte e sul Residual Feed Intake di 451 vacche da latte allevate negli Stati Uniti ed in Canada. Gli stessi autori hanno anche utilizzato il concetto di holobiability: un nuovo termine coniato da Saborío-Montero e colleghi (2021) che identifica la varianza dovuta all’azione congiunta del genoma dell’ospite (ereditabilità) e del microbiota (microbiabilità).
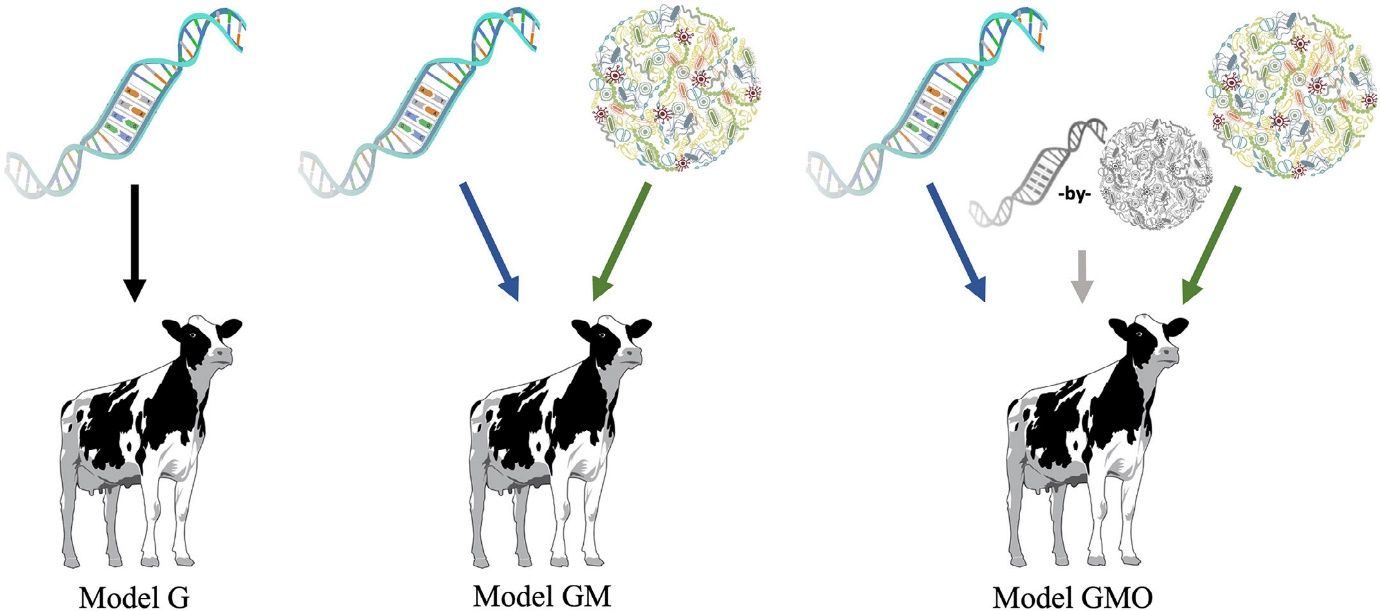
Effetto del Genoma della vacca (G), del Genoma della vacca e del Microbiota (GM) e della loro interazione (GMO). Fonte: Boggio et al https://doi.org/10.3168/jds.2023-23869
Tra i risultati più interessanti ci sono i valori di Microbiabilità che variano dal 18 al 26%, ma soprattutto quelli dell’holobiability, che varia da un minimo del 43% per il Residual Feed Intake fino ad un massimo del 62% per il valore energetico del latte. Questi risultati suggeriscono quindi che l’interazione tra il genoma dell’ospite e quello del microbiota hanno un certo peso nei caratteri dell’efficienza alimentare.
Anche negli ovini sono stati condotti dei primi studi sul rapporto tra il microbioma del latte e la sua composizione. Boggio e colleghi (2023b) hanno utilizzato dati di 795 pecore di razza Lacaune ma i loro risultati non hanno confermato quanto ottenuto nei bovini da latte, avendo stimato una microbiabilità prossima allo 0 ed avendo osservato che anche inserendo l’effetto del microbioma nel modello, la sua accuratezza non aumentava.
Infine He et al (2022) hanno valutato l’effetto del genoma dell’ospite e del microbiota di 3 diverse razze di suini (Duroc, Landrace e Large White) su alcuni caratteri di accrescimento e composizione trovando risultati di microbiabilità che variavano dal 7 al 21% per la Duroc, dal 3 al 21% per la Landrace e dal 2 al 24% per la Large White. Anche in questo caso, risultati sicuramente apprezzabili.
Considerazioni Finali
“Le parole sono i chiodi per attaccare le idee”, così diceva André Godin e non aveva torto perché molta strada è stata fatta nell’ambito di un argomento che di complesso ha anche il nome: microbiota.
Le nuove metodiche di sequenziamento ci hanno aiutato in questo e ci hanno fornito molto materiale su cui lavorare. I primi risultati sono sicuramente interessanti ma, sebbene promettenti, sono in parte contrastanti perché sembrano essere specie-specifici e molto dipendenti dalla dimensione dei dati utilizzati per ottenerli.
Non è facile poter prevedere un’applicazione diretta in campo soprattutto per i costi, ancora alti, ma in particolare per la tuttora non facile raccolta del fenotipo. Anche in questo caso dipende dalla specie, perché ad esempio nei suini o nei polli, che hanno strutture di breeding molto centralizzate, la raccolta del microbiota è sicuramente più semplice che non nelle stalle da latte ‘commerciali’.
Ad ogni modo ci si sta muovendo anche su questo e, come è successo per la genomica, gli investimenti e la ricerca non tarderanno a rendere il microbiota uno nuovo compagno di viaggio.
Bibliografia
Boggio, G. M., Monteiro, H. F., Lima, F. S., Figueiredo, C. C., Bisinotto, R. S., Santos, J. E., … & Peñagaricano, F. (2023a). Host and rumen microbiome contributions to feed efficiency traits in Holstein cows. Journal of Dairy Science.
Boggio, G. M., Christensen, O. F., Legarra, A., Meynadier, A., & Marie-Etancelin, C. (2023b). Microbiability of milk composition and genetic control of microbiota effects in sheep. Journal of Dairy Science, 106(9), 6288-6298.
Difford, G., Lassen, J., & Lovendahl, P. (2016). Genes and microbes, the next step in dairy cattle breeding. In Book of Abstracts of the 67th Annual Meeting of the European Federation of Animal Science (No. 22, pp. 285-285)
Difford, G. F., Plichta, D. R., Løvendahl, P., Lassen, J., Noel, S. J., Højberg, O., … & Sahana, G. (2018). Host genetics and the rumen microbiome jointly associate with methane emissions in dairy cows. PLoS genetics, 14(10), e1007580.
He, Y., Tiezzi, F., Jiang, J., Howard, J., Huang, Y., Gray, K., … & Maltecca, C. (2022). Exploring methods to summarize gut microbiota composition for microbiability estimation and phenotypic prediction in swine. Journal of Animal Science, 100(9), skac231.
Hou, J., Xiang, J., Li, D., Liu, X., & Pan, W. (2022). Gut microbial response to host metabolic phenotypes. Frontiers in Nutrition, 9, 1019430.
Matthews, C., Crispie, F., Lewis, E., Reid, M., O’Toole, P. W., & Cotter, P. D. (2019). The rumen microbiome: a crucial consideration when optimising milk and meat production and nitrogen utilisation efficiency. Gut microbes, 10(2), 115-132.
Ross EM, Moate PJ, Bath CR, Davidson SE, Sawbridge TI, Guthridge KM, et al. High throughput whole rumen metagenome profiling using untargeted massively parallel sequencing. BMC Genet [Internet]. BMC Genetics; 2012;13(1):53. Available from: BMC Genetics
Saborío-Montero, Alejandro, Mónica Gutiérrez-Rivas, Adrián López-García, Aser García-Rodríguez, Raquel Atxaerandio, Idoia Goiri, José Antonio Jiménez-Montero, and Oscar González-Recio. “Holobiont effect accounts for more methane emission variance than the additive and microbiome effects on dairy cattle.” Livestock Science 250 (2021): 104538.
















































































