Sappiamo bene quanto l’ambiente influenzi la produzione, la riproduzione e il benessere degli animali da reddito. In realtà, però, l’impatto che un certo ambiente, o le sue variazioni, può avere su ogni individuo non è lo stesso, e questo vuol dire che la risposta di due individui a due diversi ambienti può essere completamente diversa, sia come direzione che intensità. Un aspetto questo che, anche per la sua complessità, solo in parte è oggi considerato nelle valutazioni genetiche. Vediamo quali opzioni metodologiche sono disponibili per poter gestire questo importante rapporto.
Nel suo libro Atomic Habits, lo scrittore James Clear afferma che l’ambiente che ci circonda è la mano invisibile che modella il nostro comportamento. In effetti, se torniamo nel nostro settore e pensiamo agli animali da reddito, sappiamo bene che l’ambiente ha un grande impatto sulle loro performance – basti pensare ai caratteri della salute.
Non solo, sappiamo anche che esiste una componente genetica che ci interessa per fare selezione e che si somma all’effetto dell’ambiente. C’è però anche “la mano invisibile” di cui parla Clear, che nel nostro caso si materializza in quell’interazione genotipo-ambiente che porta ad una diversa risposta di due individui a due diversi ambienti, sia in termini di direzione che di intensità.
L’interazione Genotipo-Ambiente (GxE)
Il concetto di GxE non è un argomento recente perché già agli inizi del secolo scorso i lavori pionieri di Haldane (1946), Hammond (1947) e Falconer (1952) avevano iniziato ad introdurre la problematica suggerendo anche metodi per poterla gestire. Di sicuro è un aspetto complesso, e questo ha fatto sì che negli anni i principali modelli utilizzati per le valutazioni genetiche non lo considerassero direttamente se non, come vedremo a breve, con approcci multicarattere.
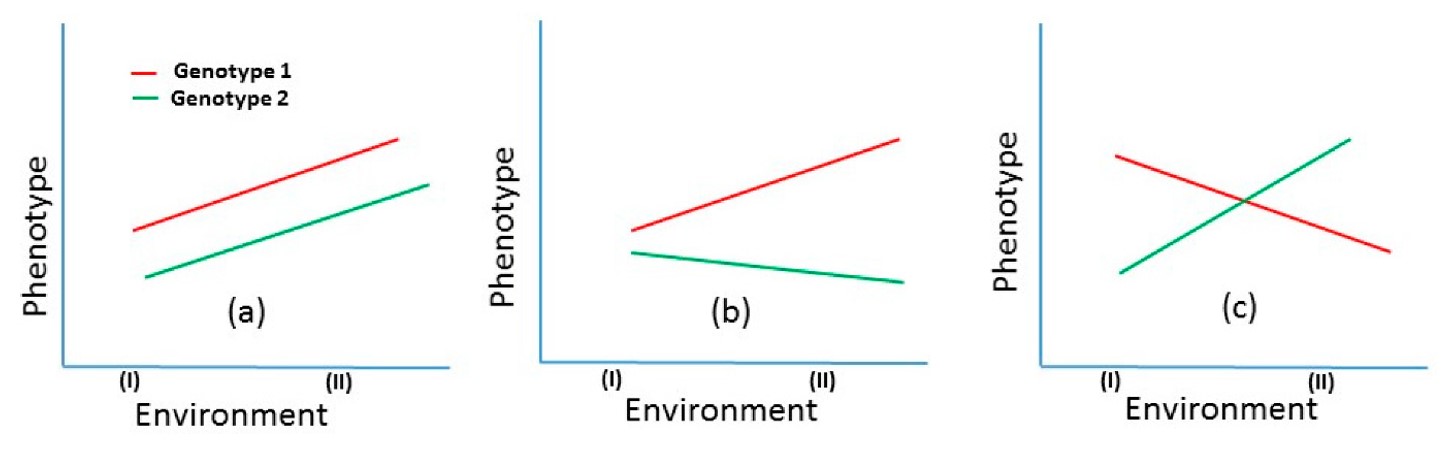
In estrema sintesi il concetto di GxE può essere facilmente colto osservando la Figura 1, tratta da un lavoro di Hasan et al (2024).
Nei riquadri a, b e c è rappresentata la risposta di due genotipi diversi (1 e 2) a due diversi ambienti (I e II). Nel riquadro a, i due genotipi hanno la stessa risposta, sia come direzione che intensità, e possiamo quindi affermare che l’interazione GxE è assente.
Nel riquadro b, invece, notiamo qualcosa di diverso: entrambi i genotipi cambiano la loro risposta. Nell’ambiente I sono molto simili mentre nel II le loro differenze si acuiscono, favorendo il genotipo 1. Siamo quindi di fronte ad una situazione di interazione GxE, anche se non estrema.
Infine, nel riquadro c possiamo osservare la situazione più estrema di GxE, dove il genotipo 1 si comporta meglio nell’ambiente I e il genotipo 2 nell’ambiente II.
Dal punto di vista della selezione questi diversi scenari possono avere un impatto non trascurabile. Esistono però degli approcci che possono essere utilizzati per gestire queste situazioni: i modelli multicarattere ed i modelli così detti reaction norm, che la traduzione italiana “a norma di reazione” rende meno affascinanti.
Il modello multicarattere per la valutazione del GxE
Pensiamo alle figlie di tori diversi allevate in paesi o regioni diverse: che facciano latte o carne è molto probabile che le loro performance siano influenzate dall’ambiente in modo diverso – c’è quindi interazione GxE. In una situazione di questo tipo possiamo considerare il fenotipo latte osservato nel paese A come un carattere diverso dal fenotipo latte osservato nel paese B.
Questo può sembrare inizialmente un nonsenso perché il latte è latte ma in realtà non lo è perché i due caratteri latte A e latte B sono legati tra di loro da una correlazione genetica più o meno forte, ad indicare con questo un effetto più o meno forte dell’interazione GxE.
Se la correlazione genetica tra i 2 caratteri è pari a 1 vuol dire che non c’è GxE ed i vari genotipi si comportano allo stesso modo in ambienti diversi.
Quando le correlazioni genetiche sono inferiori ad 1 allora siamo in presenza di GxE e la risposta dei vari genotipi non è la stessa nei diversi ambienti. Questo tipo di approccio è quello utilizzato da ormai oltre 20 anni da Interbull per le valutazioni internazionali dei Bovini da latte e più recentemente da Interbeef per quanto riguarda i bovini da carne.
A titolo di esempio, utilizzando le informazioni fornite da Interbull (e disponibili a questo link), possiamo verificare che la correlazione genetica del carattere latte nella Frisona Italiana varia da un minimo di 0.64 con la Croazia ad un massimo di 0.90 con gli Stati Uniti. Questo vuol dire che uno stesso toro si comporterà – geneticamente – in modo estremamente diverso in Croazia rispetto all’Italia, mentre tra Italia e USA la risposta del genotipo ai due ambienti sarà molto più simile.
L’interazione GxE con i modelli reaction norm
L’esempio precedente partiva dal presupposto che le figlie di uno stesso toro fossero allevate in ambienti fisicamente molto distanti/diversi tra loro, come ad esempio due nazioni diverse. Anche due stalle separate da qualche kilometro sono ambienti diversi ma non così distinti da ipotizzare un approccio multicarattere – vorrebbe dire avere migliaia di caratteri latte.
In una situazione come questa, l’effetto del diverso ambiente su un genotipo può essere però identificato o quantificato da una qualche variabile continua, come ad esempio il THI, oppure il tenore di sostanza secca nella razione, l’andamento dell’età al primo parto o l’andamento della gestione aziendale, insomma da tutta una serie di variabili che presentano un gradiente il cui variare influenza la risposta del genotipo stesso.
Si parla di “norma di reazione” perché si osserva il cambiamento (continuo) nell’espressione di un genotipo (la sua reazione) al variare delle condizioni ambientali.
Il problema di questi modelli è che sono un pò più complessi da interpretare e si basano, solitamente, su approcci di regressione lineare. Come in una regressione lineare ci sono quindi 2 parametri che quantificano la variabilità osservata, in questo caso la reazione di un genotipo all’ambiente.
Il primo, l’intercetta, definisce il contributo genetico che non dipende dall’ambiente, mentre il secondo, la pendenza, è il vero indicatore di GxE.
La Figura 2, tratta da Manuck (2010), cerca di sintetizzare questo concetto confrontando la reazione di 3 genotipi a, b e c ad un gradiente ambientale continuo.
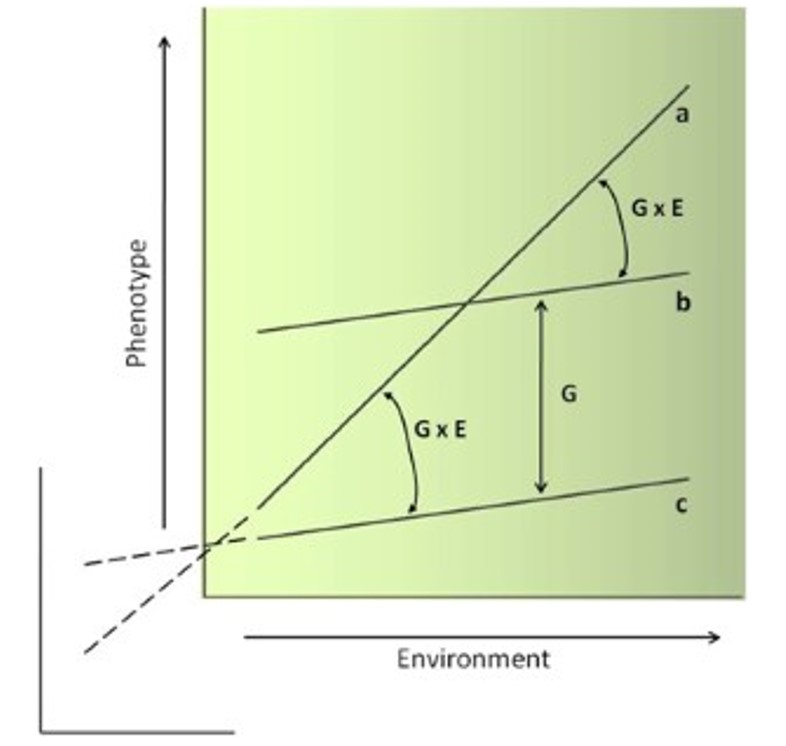
Figura 2. Esempio di Reaction Norms di 3 genotipi a, b e c. Tratto da Manuck, S (2010). https://doi.org/10.1038/mp.2009.139
I due genotipi b e c reagiscono al gradiente ambientale nello stesso modo, ed infatti la pendenza della loro risposta rimane la stessa sebbene con livelli genetici diversi (intercetta della reaction norm per ciascun genotipo b e c quantificata dalla differenza G).
Il genotipo a è invece quello che reagisce al gradiente ambientale in modo completamente diverso, sia rispetto al genotipo c che a quello b, con una risposta che è funzione del gradiente ambientale.
Nonostante la iniziale complessità, questo tipo di approccio sta diventando quello più utilizzato in diverse specie e diversi caratteri, anche e soprattutto con l’aggiunta delle informazioni genomiche. Esempi più o meno recenti li troviamo nei bovini da latte per il carattere riproduzione (Liu et al, 2019, Shi et al 2021), in quelli da carne per l’efficienza alimentare (Silva Neto et al, 2023), e nei suini per l’effetto dello stress termico nei maiali (Freitas et al, 2024).
Considerazioni finali
Tra le varie conseguenze dei mutamenti ambientali c’è sicuramente il loro impatto sulle performance degli animali da reddito. Questo sta cambiando anche i paradigmi con i quali si sviluppano e si applicano i modelli di valutazione genetica, in particolare per renderli più efficienti, informativi e aderenti alla realtà.
Uno dei paradigmi è quello della gestione dell’effetto interazione genotipo-ambiente. È indubbio che la componente di varianza dovuta a questa interazione non sia banale e per questo motivo debba essere correttamente identificata e quantificata.
Oggi ci sono anche le informazioni genomiche che ci permettono di quantificare con maggiore precisione come e quanto i diversi individui rispondono a gradienti ambientali diversi.
Un esempio per tutti potrebbe essere l’interazione genotipo-ambiente nel caso della produzione di metano, un argomento veramente “caldo”.
Bibliografia consultata
- Haldane JB. The interaction of nature and nurture. Annals of eugenics. 1946 Jan;13(1):197-205.
- Hammond J. Animal breeding in relation to nutrition and environmental conditions. Biological Reviews. 1947 Jul;22(3):195-213.
- Hasan, M.M.; Thomson, P.C.; Raadsma, H.W.; Khatkar, M.S. A Review and Meta-Analysis of Genotype by Environment Interaction in Commercial Shrimp Breeding. Genes 2024, 15, 1222. https://doi.org/10.3390/genes15091222
- Falconer DS. The problem of environment and selection. The American Naturalist. 1952 Sep 1;86(830):293-8.
- Freitas PH, Johnson JS, Tiezzi F, Huang Y, Schinckel AP, Brito LF. Genomic predictions and GWAS for heat tolerance in pigs based on reaction norm models with performance records and data from public weather stations considering alternative temperature thresholds. Journal of Animal Breeding and Genetics. 2024 May;141(3):257-77.
- Liu A, Su G, Höglund J, Zhang Z, Thomasen J, Christiansen I, Wang Y, Kargo M. Genotype by environment interaction for female fertility traits under conventional and organic production systems in Danish Holsteins. Journal of Dairy science. 2019 Sep 1;102(9):8134-47.
- Manuck SB. The reaction norm in gene x environment interaction. Molecular Psychiatry. 2010 Sep;15(9):881-2.
- Shi R, Brito LF, Liu A, Luo H, Chen Z, Liu L, Guo G, Mulder H, Ducro B, van der Linden A, Wang Y. Genotype-by-environment interaction in Holstein heifer fertility traits using single-step genomic reaction norm models. BMC genomics. 2021 Dec;22:1-20.
- Silva Neto JB, Mota LF, Amorim ST, Peripolli E, Brito LF, Magnabosco CU, Baldi F. Genotype-by-environment interactions for feed efficiency traits in Nellore cattle based on bi-trait reaction norm models. Genetics Selection Evolution. 2023 Dec 14;55(1):93.








Scrivi un commento
Devi accedere, per commentare.